Как проверить занятость домена? Что делать, если нужный домен занят?
Выбирая новый домен, самое первое, что нужно сделать – проверить имя домена на занятость. Если вы придумали оригинальное название своему сайту, вероятнее всего, оно уже занято. Можно просто проверить занятость интересующих слов или словосочетаний. В основных зонах все простые и красивые имена давно расхватаны, поэтому прежде, чем найти домен для своего ресурса, приходится перебрать несколько десятков вариантов. Итак, как проверить домен на занятость?
Простейший способ
Простой, быстрый способ проверить занят ли домен – вписать нужное вам имя в поисковую строку браузера и нажать ввод. Если браузер откроет какую-либо страницу, значит, домен занят. Он будет занят даже в том случае, если страница «парковочная», то есть выставленная на продажу. Чтобы приобрести это имя, нужно будет связаться с его владельцем. Цена при этом будет намного выше стоимости стандартной регистрации.
Несмотря на то, что этот способ проверить занят ли домен признан самым легким (не требует использования никаких сервисов), стопроцентно надежным он не считается. Прибегать к нему можно в самом начале, чтобы выбрать и отсеять занятые имена. Более высокую точность проверки гарантируют специальные сервисы whois.
Что такое сервисы whois?
Сервисы whois изначально были созданы для уточнения данных о владельце ресурса. Теперь их используют для проверки регистрации домена. Таких сервисов сейчас очень много, но пользоваться малоизвестными не рекомендуется. Некоторые владельцы непопулярных сервисов whois могут сохранять поисковые запросы. В случае если вы придумаете красивое имя вашему сайту и оно окажется свободным, лучше сразу его зарегистрировать. Иначе, «засветив» имя, вы сделаете подарок владельцу whois.
Поэтому проверить занят ли домен на занятость лучше у авторитетных и крупных регистраторов. Это более надежно и безопасно. Даже при существующем сохранении запросов они не будут заниматься регистрацией найденного вами домена, так как это не представляет интереса.
Все, что вам нужно – это ввести имя домена в поисковую строку сервиса whois и нажать кнопку Search. После чего программа выдаст вам данные о владельце домена, о регистраторе и технической поддержке. Если имя окажется свободным, не медлите и приступайте к регистрации.
Что делать, если нужный домен занят?
Самый простой вариант – найти другое имя. Но если делать этого не хочется, то его можно просто купить. Если домен занят, значит, на нем есть какой-либо сайт. Воспользуйтесь обратной связью и сообщите владельцу, что намереваетесь приобрести его домен. Если это всего лишь персональная страница с низкой посещаемостью, то у вас имеются все шансы заполучить имя. Причем сделка окажется выгодной – вы купите домен по себестоимости или за пару сотен долларов.
Если вы попали на портал с высокой посещаемостью – скорее всего, вам придется забыть выбранное имя!
Часто случается, что красивое имя домена занято, но его не используют. Это значит, что владелец планирует продать его. Нужно связаться с ним и договориться о цене. Стоимость домена варьирует от нескольких сотен до нескольких тысяч долларов. Она напрямую зависит от самого имени и от зоны расположения. В среднем приемлемая цена хорошего дома составляет от 500 до 1 500 долларов. Все сделки подобного рода совершаются с доменами com или ru.
Нужно связаться с ним и договориться о цене. Стоимость домена варьирует от нескольких сотен до нескольких тысяч долларов. Она напрямую зависит от самого имени и от зоны расположения. В среднем приемлемая цена хорошего дома составляет от 500 до 1 500 долларов. Все сделки подобного рода совершаются с доменами com или ru.
Не стоит претендовать на имена доменов, зарегистрированные на владельцев известных торговых марок. Это заведомо проигрышное дело.
Во избежание лишних проблем и траты времени лучшим вариантом будет самостоятельно в интересующей вас зоне выбрать подходящее имя.
Узнайте также о плюсах и минусах цифровых доменов.
Предложить идею урока:
Как проверить домен на занятость?
Главная » Обратите внимание
Обратите внимание
Автор sorc39 На чтение 3 мин. Просмотров 1 Опубликовано
Просмотров 1 Опубликовано
Наверняка каждый из нас в тот или иной период своей жизни задумывался о создании сайта на ту или иную тематику. Копнув поглубже в тему все мы сталкивались с неизбежностью регистрации домена.
Чтобы проверить домен на занятость, требуется воспользоваться специальным сервисом. Он проверяет доступность его регистрации. Но чтобы была возможность им воспользоваться, нужно сначала вашему сайту придумать имя. Обычно пользователи подбирают какое-то слово, которое способно охарактеризовать тематику будущего ресурса.
Как проверить?
- Сейчас в интернете есть большое число ресурсов, на которых есть возможность за короткое время проверить в популярных зонах наличие свободного домена. Если вы не знаете, где будете покупать домен, то есть сайты, где можно проверить свободное имя домена. К примеру, это может быть сервис www.
 check.ru.
check.ru. - Для проверки необходимо сначала выбрать имя будущего домена и зону. Далее нажимаете кнопку поиска, и система сообщит вам о занятости домена. При проверке вам могут сразу сделать предложение приобрести данный свободный домен. Специалисты советуют покупать домен сразу после того, как он был проверен. Бывает и так, что через пару минут домен оказывается уже занятым.
- Учитывая такой фактор, сначала стоит тщательно изучить информацию о цене регистрации домена у какой-то компании в определенных зонах. Более того, у вас будет возможность зарегистрировать его по привлекательной для вас цене.
Сейчас, если взять зону ru, то здесь заняты практически все однословные домены. Поэтому вам придется использовать приставку или выбирать несколько доменных зон.
На сайте, где вы будете проверять домен, вам предложат ввести в определенную форму тот домен, который вы придумали. Потом нужно поставить галочки в необходимых доменных зонах и нажать кнопку «Проверить».
Что делать, если ваш домен занят?
Если же ваш домен занят, то можно воспользоваться специальными приставками. Любая поисковая система имеет собственный алгоритм ангажирования. К примеру, у Яндекса – это ТИЦ, у других могут быть другие.
Бывает и так что вам приходится регистрировать тот домен, которым кто-то уже пользовался. В таком случае есть большой плюс. У этого домена не будет нулевых показателей, так как их рост зависит от того, сколько будет ссылаться сайтов на ваш.
Сегодня создать свой сайт можно за несколько минут. Многие пользователи, мечтающие о собственном интернет-пространстве, могут осуществить свои мечты. Каждый сайт имеет определенный адрес. После проверки доменного имени, есть возможность его зарегистрировать.
Онлайн-адаптация домена для картирования занятости · Робототехника: наука и системы
Онлайн-адаптация домена для картирования занятости
Энтони Томпкинс, Рансалу Сенанаяке, Фабио Рамос
Abstract
Создание точных пространственных представлений с учетом неопределенности имеет решающее значение для безопасной навигации автономных роботов в неструктурированных средах.

Live Paper Discussion Information
| Start Time | End Time | |
|---|---|---|
| 07/16 15:00 UTC | 07/16 17:00 UTC |
Virtual Conference Presentation
Дополнительное видео
Обзоры статей
Обзор 1 Я считаю, что эта статья очень интересна, а общие идеи достойны публикации. Постановка задачи обещает быть полезной для многих приложений, а вычислительная нагрузка (которая рассматривается в данной работе) в этом классе подходов в настоящее время является одним из основных ограничивающих факторов, препятствующих широкому внедрению. Меня больше всего беспокоит нынешнее состояние работы — ее понятность и оценки.
Что касается понятности, без знания предшествующей работы в этой области, настоящую статью относительно сложно разобрать. Хотя гл. II-a значительно помогает, знание [13] или одной из статей о картах Гильберта, по-видимому, все еще требуется. Кроме того, я считаю, что в работе следует более четко/ранее указать, что задачи отображения в новых средах рассматриваются как задача адаптации предметной области.
Во-вторых, оценка рассматривает в основном другие подходы аналогичного типа. Однако, поскольку заявленная авторами цель этой работы состоит в преодолении ограничений для реального использования, было бы интересно посмотреть, как работает предлагаемый подход по сравнению с методами лидарного картирования, которые в настоящее время используются на практике.
Постановка задачи обещает быть полезной для многих приложений, а вычислительная нагрузка (которая рассматривается в данной работе) в этом классе подходов в настоящее время является одним из основных ограничивающих факторов, препятствующих широкому внедрению. Меня больше всего беспокоит нынешнее состояние работы — ее понятность и оценки.
Что касается понятности, без знания предшествующей работы в этой области, настоящую статью относительно сложно разобрать. Хотя гл. II-a значительно помогает, знание [13] или одной из статей о картах Гильберта, по-видимому, все еще требуется. Кроме того, я считаю, что в работе следует более четко/ранее указать, что задачи отображения в новых средах рассматриваются как задача адаптации предметной области.
Во-вторых, оценка рассматривает в основном другие подходы аналогичного типа. Однако, поскольку заявленная авторами цель этой работы состоит в преодолении ограничений для реального использования, было бы интересно посмотреть, как работает предлагаемый подход по сравнению с методами лидарного картирования, которые в настоящее время используются на практике.

Документ основан на существующей работе (Automorphing Bayesian Hilbert Maps, или ABHM) для создания карт заполняемости. Поскольку вычислительная сложность ABHM не позволяет масштабировать его до сценариев реального времени, в статье предлагается оптимальная передача параметров. Передаточный параметр или матрица связи могут быть вычислены практически в режиме реального времени, что является значительным улучшением по сравнению с вычислением параметров байесовской модели. В целом предлагаемый подход представляет собой разумную комбинацию существующих методов. В документе четко описывается проблема и хорошо мотивируется использование оптимального переноса параметров.
Техническое качество бумаги нуждается в некоторых улучшениях. Хотя мотивация передачи параметров ясна, сомнительно, что предлагаемый подход дает хорошие результаты или нет. Вычисление NLL может дать нам некоторое представление об этом, но я не уверен, насколько хорошо передача параметров работает в реальных реальных сценариях.

Белковый ландшафт бактериального генома
- Список журналов
- Рукописи авторов HHS
- PMC2763621
молярная ячейка. Авторская рукопись; доступно в PMC 2010 31 июля.
Опубликовано в окончательной редакции как:
Мол. ячейка. 2009 г., 31 июля; 35(2): 247–253.
DOI: 10.1016/j.molcel.2009.06.035
PMCID: PMC2763621
NIHMSID: NIHMS133392
PMID: 19647521
Информация Автор Копа фундаментальный для основных биологических процессов, включая транскрипцию, репликацию ДНК и хромосомную организацию. Мы разработали In vivo Protein Occupancy Display (IPOD) — технология, которая показывает присутствие белка во всей бактериальной хромосоме с разрешением отдельных участков связывания. Применение к Escherichia coli выявляет тысячи пиков занятости белка, сильно обогащенных внутри и в непосредственной близости от некодирующих регуляторных областей. Кроме того, мы обнаружили обширные (> 1 килобаз) домены занятости белка (EPOD), некоторые из которых локализованы в генах с высокой экспрессией, обогащенных РНК-полимеразой. Однако большинство локализовано в транскрипционно-молчащих локусах, в которых преобладают консервативные гипотетические ORF. Эти области сильно обогащены как предсказанными, так и экспериментально определенными сайтами связывания нуклеоидных белков и демонстрируют экстремальные биофизические характеристики, такие как высокая внутренняя кривизна. Наши наблюдения указывают на то, что эти транскрипционно-молчащие EPODs являются неуловимыми организующими центрами, давно предполагаемыми для топологической изоляции хромосомных доменов.
Мы разработали In vivo Protein Occupancy Display (IPOD) — технология, которая показывает присутствие белка во всей бактериальной хромосоме с разрешением отдельных участков связывания. Применение к Escherichia coli выявляет тысячи пиков занятости белка, сильно обогащенных внутри и в непосредственной близости от некодирующих регуляторных областей. Кроме того, мы обнаружили обширные (> 1 килобаз) домены занятости белка (EPOD), некоторые из которых локализованы в генах с высокой экспрессией, обогащенных РНК-полимеразой. Однако большинство локализовано в транскрипционно-молчащих локусах, в которых преобладают консервативные гипотетические ORF. Эти области сильно обогащены как предсказанными, так и экспериментально определенными сайтами связывания нуклеоидных белков и демонстрируют экстремальные биофизические характеристики, такие как высокая внутренняя кривизна. Наши наблюдения указывают на то, что эти транскрипционно-молчащие EPODs являются неуловимыми организующими центрами, давно предполагаемыми для топологической изоляции хромосомных доменов.
Ключевые слова: IPOD, взаимодействие белок-ДНК, транскрипционные сети, нуклеоидные белки, хромосомная организация, E. coli
Репликация, поддержание и экспрессия генетической информации — это процессы, которые организованы посредством точного взаимодействия сотен белков. с хромосомной ДНК. В течение десятилетий исследования были сосредоточены на поведении и функциональных последствиях ДНК-белковых взаимодействий в отдельных локусах. Однако понимание поведения на системном уровне, такого как организация хромосом, репликация генома и динамика транскрипционной сети, требует наблюдений в масштабе всей системы. Иммунопреципитация хроматина на основе микрочипов (ChIP-chip) позволяет проводить глобальные измерения хромосомной занятости для отдельных белков (Ren et al., 2000). В другом глобальном подходе, защите от метилазы, контролируется часть всех занятых сайтов in vivo , независимо от идентичности связанных белков (Tavazoie and Church, 1998). Однако в настоящее время не существует комплексного подхода для одновременного мониторинга с высоким разрешением всех in vivo взаимодействий белок-ДНК в геноме. Мы разработали такую технологию и использовали ее для профилирования заполнения белками хромосомы E. coli при разрешении отдельных сайтов связывания.
Однако в настоящее время не существует комплексного подхода для одновременного мониторинга с высоким разрешением всех in vivo взаимодействий белок-ДНК в геноме. Мы разработали такую технологию и использовали ее для профилирования заполнения белками хромосомы E. coli при разрешении отдельных сайтов связывания.
Чтобы глобально профилировать занятость всех белков на хромосомной ДНК, мы сначала стабилизировали in vivo взаимодействий белок-ДНК посредством ковалентного перекрестного связывания с формальдегидом (). После лизиса клеток и обработки ультразвуком белковые следы минимизируются до режима ~ 50 п.н. за счет расщепления ДНКазой I (). Затем экстракцию фенолом используют для улавливания амфипатических комплексов белок-ДНК на границе раздела между органической и водной фазами. После изоляции интерфейса и реверсирования поперечных связей короткие фрагменты ДНК помечают концами и гибридизуют с мозаичной матрицей высокой плотности, содержащей 25-мерные олигонуклеотиды с разрешением одна каждые четыре пары оснований во всем геноме. После сканирования и нормализации данных достигается глобальный профиль занятости белка с высоким разрешением. Для каждого зонда на чипе уровни обогащения или истощения белка количественно оцениваются с использованием z — показатель, который представляет собой относительную интенсивность сигнала от зонда к зонду по отношению к среднему и нормализованному к стандартному отклонению сигналов от повторных гибридизаций цельной геномной ДНК ( Методы ).
После сканирования и нормализации данных достигается глобальный профиль занятости белка с высоким разрешением. Для каждого зонда на чипе уровни обогащения или истощения белка количественно оцениваются с использованием z — показатель, который представляет собой относительную интенсивность сигнала от зонда к зонду по отношению к среднему и нормализованному к стандартному отклонению сигналов от повторных гибридизаций цельной геномной ДНК ( Методы ).
Открыть в отдельном окне
In vivo отображение занятости белка (IPOD)
(A) Схема выделения и полногеномного отображения участков, связанных с белком, в бактериальном геноме. Сшивающие формальдегид консерванты in vivo взаимодействий белок-ДНК. После лизиса клеток и обработки ультразвуком белковые следы сводятся к минимуму за счет обработки ДНКазой I. Экстракция фенолом обогащает комплексы белок-ДНК на границе раздела водной и органической фаз.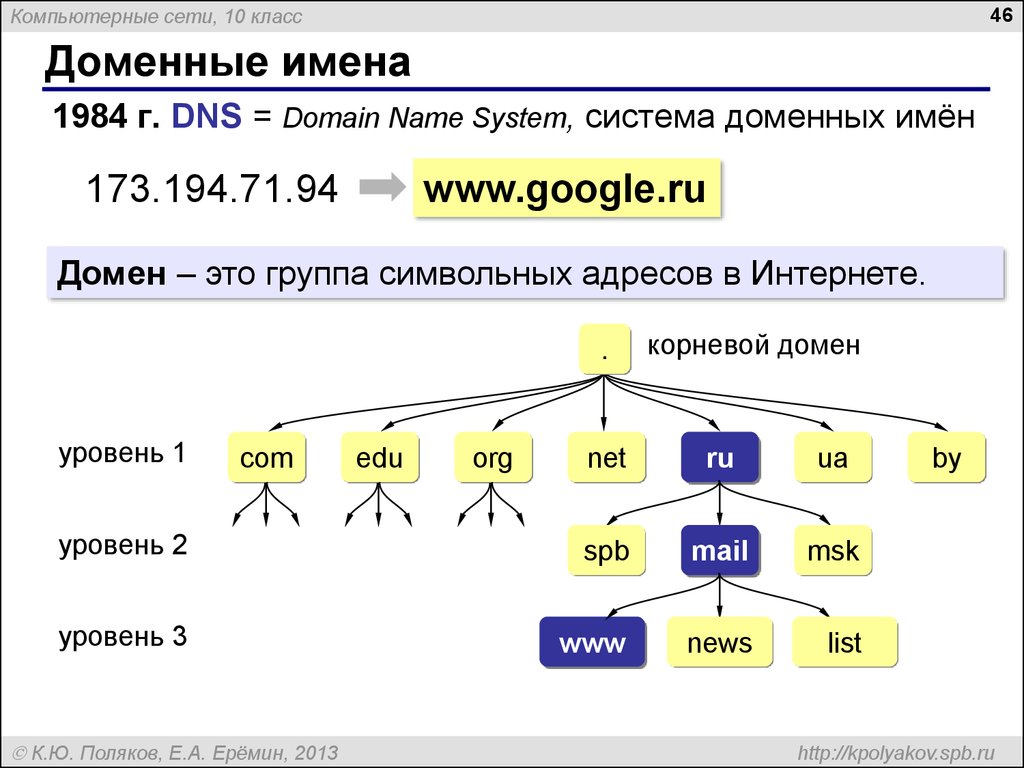 После выделения интерфейса перекрестные связи меняют местами, полученные фрагменты ДНК метят на концах и гибридизуют с массивом плиток. (B) Гель-фракционирование показывает, что обработка ДНКазой I приводит к падению режима распределения длин фрагментов с ~1000 п.н. (без ДНКазы I) до ~200 п.н. (½X ДНКазы I), до менее 100 п.н. для ( 1X ДНКаза I). Образцы разделяли на одном и том же геле и удаляли посторонние полосы для ясности. (C) Кумулятивное распределение вероятностей занятости ( z -балл: стандартное отклонение от среднего) как для кодирующих, так и для некодирующих областей, определенное во время поздней экспоненциальной фазы роста. Значения z баллов были сглажены путем усреднения в скользящем окне из 128 пар оснований.
После выделения интерфейса перекрестные связи меняют местами, полученные фрагменты ДНК метят на концах и гибридизуют с массивом плиток. (B) Гель-фракционирование показывает, что обработка ДНКазой I приводит к падению режима распределения длин фрагментов с ~1000 п.н. (без ДНКазы I) до ~200 п.н. (½X ДНКазы I), до менее 100 п.н. для ( 1X ДНКаза I). Образцы разделяли на одном и том же геле и удаляли посторонние полосы для ясности. (C) Кумулятивное распределение вероятностей занятости ( z -балл: стандартное отклонение от среднего) как для кодирующих, так и для некодирующих областей, определенное во время поздней экспоненциальной фазы роста. Значения z баллов были сглажены путем усреднения в скользящем окне из 128 пар оснований.
Глобальный профиль заполнения белками хромосомы
E. coli Большая часть охарактеризованных взаимодействий белок-ДНК происходит посредством специфичных для последовательности взаимодействий факторов транскрипции с ДНК внутри и в непосредственной близости от некодирующих регуляторных областей ( Гама-Кастро и др. , 2008). В соответствии с этим мы видим очень значительное обогащение занятости в некодирующих областях по сравнению с кодирующими областями (). Эта разница в размещении ясно различима в локальном хромосомном представлении, где высокоамплитудные пики в основном ограничены областями между генами (4). Независимые биологические повторы демонстрируют, что положение и относительная амплитуда этих пиков заполнения показывают высокий уровень воспроизводимости (1). Хотя в целом наблюдается относительное истощение занятости в пределах открытых рамок считывания (ORF), иногда это прерывается резким пиком занятости (, S1). Функциональная роль этих внутригенных взаимодействий неизвестна, но может представлять собой значительный пробел в нашем понимании экспрессии бактериальных генов. При высоком разрешении можно легко различить заселенность отдельных белков, отображая следы в масштабе типичного сайта связывания фактора транскрипции (, S2). Алгоритм автоматического обнаружения пиков идентифицировал ~ 2063 отдельных занятых сайта в популяции из 9 человек.
, 2008). В соответствии с этим мы видим очень значительное обогащение занятости в некодирующих областях по сравнению с кодирующими областями (). Эта разница в размещении ясно различима в локальном хромосомном представлении, где высокоамплитудные пики в основном ограничены областями между генами (4). Независимые биологические повторы демонстрируют, что положение и относительная амплитуда этих пиков заполнения показывают высокий уровень воспроизводимости (1). Хотя в целом наблюдается относительное истощение занятости в пределах открытых рамок считывания (ORF), иногда это прерывается резким пиком занятости (, S1). Функциональная роль этих внутригенных взаимодействий неизвестна, но может представлять собой значительный пробел в нашем понимании экспрессии бактериальных генов. При высоком разрешении можно легко различить заселенность отдельных белков, отображая следы в масштабе типичного сайта связывания фактора транскрипции (, S2). Алгоритм автоматического обнаружения пиков идентифицировал ~ 2063 отдельных занятых сайта в популяции из 9 человек. 0006 клеток E. coli , растущих в поздней экспоненциальной фазе (рис. S3). Характер пиков воспроизводим в биологических повторах и показывает вариации, зависящие от условий (рис. S4).
0006 клеток E. coli , растущих в поздней экспоненциальной фазе (рис. S3). Характер пиков воспроизводим в биологических повторах и показывает вариации, зависящие от условий (рис. S4).
Открыть в отдельном окне
Профиль занятости белка генома E. coli во время поздней экспоненциальной фазы роста некодирующие) области генома (красные стрелки). Однако подобные пики реже можно увидеть и в кодирующих областях (синие стрелки). Две независимые биологические реплики показывают хорошо воспроизводимые профили занятости в этом регионе. (B) При высоком пространственном разрешении множественные пики занятости различимы в пределах одной межгенной области. Пики локализованы на типичном следе отдельных факторов транскрипции и часто перекрываются с экспериментально определенными сайтами связывания (PurR, RegulonDB).
Открытие расширенных доменов занятости белка (EPOD)
Любопытно, что изучение профиля занятости всего генома выявило смежные области связывания белка, многие из которых превышают в длину килобазию (, S5).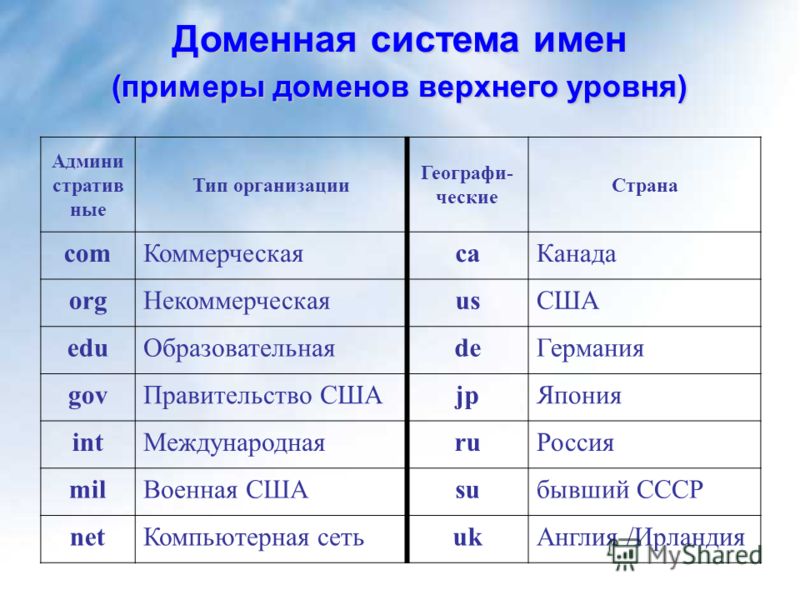 Мы выполнили систематический поиск этих расширенных доменов занятости белка (EPOD) при раннем экспоненциальном росте с использованием автоматизированного алгоритма, который идентифицировал области размером 1024 п.н. или более с непрерывными средними значениями занятости выше 75 -й -й процентиль всех полногеномных значений. Эти домены имели среднюю длину 1,6 т.п.н. и простирались до 14 т.п.н. (рис. S6A). Мы задались вопросом, соответствует ли экстремальный сигнал в этих доменах следу РНК-полимеразы в высоко транскрибируемых областях. Чтобы проверить эту возможность, мы выполнили профилирование транскрипции в идентичных условиях клеточного роста ( Methods ). Как видно (), мы обнаружили четкие случаи, когда границы EPOD совпадали с границами высоко транскрибируемых областей, таких как те, которые содержат гены рибосомных белков (). Однако мы обнаружили много случаев, когда EPOD существовали в транскрипционно-молчащем состоянии как в генах, так и в межгенных регионах и даже в длинных оперонах (, S7).
Мы выполнили систематический поиск этих расширенных доменов занятости белка (EPOD) при раннем экспоненциальном росте с использованием автоматизированного алгоритма, который идентифицировал области размером 1024 п.н. или более с непрерывными средними значениями занятости выше 75 -й -й процентиль всех полногеномных значений. Эти домены имели среднюю длину 1,6 т.п.н. и простирались до 14 т.п.н. (рис. S6A). Мы задались вопросом, соответствует ли экстремальный сигнал в этих доменах следу РНК-полимеразы в высоко транскрибируемых областях. Чтобы проверить эту возможность, мы выполнили профилирование транскрипции в идентичных условиях клеточного роста ( Methods ). Как видно (), мы обнаружили четкие случаи, когда границы EPOD совпадали с границами высоко транскрибируемых областей, таких как те, которые содержат гены рибосомных белков (). Однако мы обнаружили много случаев, когда EPOD существовали в транскрипционно-молчащем состоянии как в генах, так и в межгенных регионах и даже в длинных оперонах (, S7). Из-за их экстремального и бимодального поведения экспрессии РНК мы выполнили автоматическую классификацию EPOD, объединив их в две популяции, используя их средний уровень экспрессии по доменам (9).0089 Методы и дополнительный набор данных 1). Это привело к 121 домену в классе с высокой экспрессией ( he EPOD) и 151 в классе транскрипционно молчащих ( ts EPOD). Ранее опубликованные данные чипа РНК-полимеразы ChIP (Grainger et al., 2005) из клеток, выращенных в идентичных условиях, позволили нам сравнить занятость РНК-полимеразой ts EPOD и he EPOD по сравнению с фоновым набором, полученным путем случайной выборки. геномные последовательности из общего распределения длин EPOD (). Как и ожидалось, he EPOD показали чрезвычайно высокие уровни занятости РНК-полимеразой ( P < 10 -246 ). Для сравнения, ts EPOD показали более низкие уровни заполнения РНК-полимеразой ( P <0,02) по сравнению с контролем.
Из-за их экстремального и бимодального поведения экспрессии РНК мы выполнили автоматическую классификацию EPOD, объединив их в две популяции, используя их средний уровень экспрессии по доменам (9).0089 Методы и дополнительный набор данных 1). Это привело к 121 домену в классе с высокой экспрессией ( he EPOD) и 151 в классе транскрипционно молчащих ( ts EPOD). Ранее опубликованные данные чипа РНК-полимеразы ChIP (Grainger et al., 2005) из клеток, выращенных в идентичных условиях, позволили нам сравнить занятость РНК-полимеразой ts EPOD и he EPOD по сравнению с фоновым набором, полученным путем случайной выборки. геномные последовательности из общего распределения длин EPOD (). Как и ожидалось, he EPOD показали чрезвычайно высокие уровни занятости РНК-полимеразой ( P < 10 -246 ). Для сравнения, ts EPOD показали более низкие уровни заполнения РНК-полимеразой ( P <0,02) по сравнению с контролем.
Открыть в отдельном окне
Расширенные домены занятости белка (EPOD)
Профили занятости белка и экспрессии РНК показаны для ранней экспоненциальной фазы роста, сглажены путем усреднения в скользящем окне из 512 пар оснований. Внизу на обеих нитях аннотированы открытые рамки считывания (ORF). Автоматически обнаруженные транскрипционно-молчащие EPOD показаны вверху. (A) Домен с высоким содержанием белка и высокой экспрессией РНК, охватывающий область с генами, кодирующими субъединицы рибосомного белка. (B) EPOD автоматически обнаруживается в транскрипционно-молчащей области с генами, кодирующими продукты биосинтеза ЛПС. Соседняя область, кодирующая 50S рибосомальные белковые субъединицы ( об/мин G и об/мин B ), показывает равный уровень занятости белком, но демонстрирует высокий уровень экспрессии РНК. (C,D) Транскрипционно-молчащие EPOD, обнаруженные в генах с неизвестной функцией, кодирующих предсказанный белок, yccE (C) и консервативный белок внутренней мембраны, yliE (D) .
Открыть в отдельном окне
Различный состав занятости и биофизические свойства в расширенных доменах занятости белка
Кумулятивное распределение различных показателей показано для транскрипционно-молчащих EPOD (красный), высокоэкспрессируемых EPOD (синий) и соответствующий элемент управления фоном (черный). Критерий суммы рангов Уилкоксона используется для определения статистической значимости наблюдаемых отклонений относительно фона. (A) Экспериментально определенная относительная занятость РНК-полимеразы. (B,C) Расчетная оценка предпочтения связывания PWM для нуклеоидного белка (H-NS) и ненуклеоидного репрессора транскрипции (LacI) с использованием относительной меры для всего генома ( z -балл). (D) Совокупное распределение частот A:T в EPOD. (E) Кумулятивное распределение предсказанных значений относительной кривизны в пределах EPOD. (F) Распределение сайтов связывания различных нуклеоидных белков и СРБ в пределах ts EPOD и he EPOD.
(F) Распределение сайтов связывания различных нуклеоидных белков и СРБ в пределах ts EPOD и he EPOD.
Чтобы лучше понять потенциальную роль EPOD, мы искали обогащение определенных функциональных категорий в генах, которые их перекрывают (таблица S1). Как и ожидалось, и EPOD были сильно обогащены процессами и путями, которые высоко экспрессируются, включая трансляцию и тРНК. Наиболее значительно обогащенные классы в пределах ts EPOD были предсказаны и гипотетические ORF с незначительно значимым обогащением профаговых и связанных с профагами генов. С другой стороны, ts EPOD в целом избегают предположительно важных генов (таблица S2). Количество ts EPOD, их явно случайное, но широкое распространение по всему геному и их обогащение внутри транскрипционно-молчащих ORF с неизвестной функцией предполагает, что они могут выполнять архитектурную роль. На самом деле существуют убедительные доказательства того, что хромосома E. coli организована в домены, служащие как хромосомной компактизации, так и топологической изоляции доменов (Postow et al., 2004). Доказательства для таких 9Организация 0006 in vivo получена в результате как генетических, так и биохимических исследований (Garcia-Russell et al., 2007; Postow et al., 2004), включая визуализацию розетоподобных структур с помощью микроскопии (Delius and Worcel, 1974b; Hinnebusch and Bendich, 1997; Петтиджон, 1996; Постоу и др., 2004). Однако вопросы формирования, состава, содержания и динамики этих доменов остаются открытыми (Bendich, 2001; Postow et al., 2004; Travers and Muskhelishvili, 2007). Исследователи утверждали, что такие домены могут быть организованы за счет связывания и взаимодействия многочисленных белков, которые в совокупности называются нуклеоидными белками (Azam and Ishihama, 19).99). Эти белки обладают характеристиками, которые хорошо подходят для этой задачи. К ним относятся высокая распространенность, низкая специфичность последовательности, склонность к искривлению ДНК и склонность к связыванию искривленной ДНК.
coli организована в домены, служащие как хромосомной компактизации, так и топологической изоляции доменов (Postow et al., 2004). Доказательства для таких 9Организация 0006 in vivo получена в результате как генетических, так и биохимических исследований (Garcia-Russell et al., 2007; Postow et al., 2004), включая визуализацию розетоподобных структур с помощью микроскопии (Delius and Worcel, 1974b; Hinnebusch and Bendich, 1997; Петтиджон, 1996; Постоу и др., 2004). Однако вопросы формирования, состава, содержания и динамики этих доменов остаются открытыми (Bendich, 2001; Postow et al., 2004; Travers and Muskhelishvili, 2007). Исследователи утверждали, что такие домены могут быть организованы за счет связывания и взаимодействия многочисленных белков, которые в совокупности называются нуклеоидными белками (Azam and Ishihama, 19).99). Эти белки обладают характеристиками, которые хорошо подходят для этой задачи. К ним относятся высокая распространенность, низкая специфичность последовательности, склонность к искривлению ДНК и склонность к связыванию искривленной ДНК. Кроме того, известно, что некоторые из этих факторов (, например, H-NS) образуют как минимум гомодимерные взаимодействия (Stella et al., 2005), и эта способность, как утверждалось ранее (Dame et al., 2000; Skoko et al. , 2006) могут позволить удаленным участкам хромосом объединяться с образованием топологически изолированных доменов. Исследования ChIP-чипов с низким разрешением в отношении известных нуклеоидных белков (Grainger et al., 2006) выявили как смещение в сторону взаимодействия в некодирующих областях, так и корреляцию со связыванием Fis и H-NS, предполагая кооперативное взаимодействие нуклеоидных белков в поддержании геномной активности. архитектура.
Кроме того, известно, что некоторые из этих факторов (, например, H-NS) образуют как минимум гомодимерные взаимодействия (Stella et al., 2005), и эта способность, как утверждалось ранее (Dame et al., 2000; Skoko et al. , 2006) могут позволить удаленным участкам хромосом объединяться с образованием топологически изолированных доменов. Исследования ChIP-чипов с низким разрешением в отношении известных нуклеоидных белков (Grainger et al., 2006) выявили как смещение в сторону взаимодействия в некодирующих областях, так и корреляцию со связыванием Fis и H-NS, предполагая кооперативное взаимодействие нуклеоидных белков в поддержании геномной активности. архитектура.
Мы искали доказательства участия нуклеоидных белков в формировании ts EPOD. Доступность вероятностных моделей специфичности последовательности в виде весовых матриц положения, PWM (Gama-Castro et al., 2008), позволила нам определить потенциал относительной занятости этих регионов посредством компьютерного анализа подмножества этих факторов: H-NS, IHF и Fis (методы ). Мы обнаружили, что действительно в популяции 90 006 ts 90 007 EPOD имеют значительно более высокие баллы PWM для всех этих нуклеоидных белков (9).0006 напр. для H-NS P < 10 −28 ). То же самое было не так для и EPOD, так как их распределение баллов ШИМ незначительно отклонялось от фона (рис. S8). Напротив, распределение баллов PWM для LacI (ненуклеоидный фактор транскрипции) показало противоположную тенденцию со значительно более низкими значениями ( P < 10 -7 ) в пределах ts EPOD (). В соответствии с предпочтением нуклеоидных белков ДНК, богатой А/Т (Cho et al., 2008; Grainger et al., 2006), мы также наблюдали сильно искаженное смещение частоты A:T: 59% в пределах ts EPOD по сравнению с 49% для фона и 50% для he EPOD ( P < 10 −30 , ). Мы также обнаружили, что ts EPOD демонстрируют экстремальные биофизические характеристики (Pedersen et al., 2000), такие как высокая кривизна ( P < 10 −24 ) и энергия суммирования ( P < 10 −34 ).
Мы обнаружили, что действительно в популяции 90 006 ts 90 007 EPOD имеют значительно более высокие баллы PWM для всех этих нуклеоидных белков (9).0006 напр. для H-NS P < 10 −28 ). То же самое было не так для и EPOD, так как их распределение баллов ШИМ незначительно отклонялось от фона (рис. S8). Напротив, распределение баллов PWM для LacI (ненуклеоидный фактор транскрипции) показало противоположную тенденцию со значительно более низкими значениями ( P < 10 -7 ) в пределах ts EPOD (). В соответствии с предпочтением нуклеоидных белков ДНК, богатой А/Т (Cho et al., 2008; Grainger et al., 2006), мы также наблюдали сильно искаженное смещение частоты A:T: 59% в пределах ts EPOD по сравнению с 49% для фона и 50% для he EPOD ( P < 10 −30 , ). Мы также обнаружили, что ts EPOD демонстрируют экстремальные биофизические характеристики (Pedersen et al., 2000), такие как высокая кривизна ( P < 10 −24 ) и энергия суммирования ( P < 10 −34 ). снова согласуется с гипотезой о том, что эти регионы представляют собой хромосомные организующие центры (рис. S9). В соответствии с нашими вычислительными анализами выше, мы увидели значительное обогащение для высокоаффинного связывания нуклеоидных белков в наших ts EPOD относительно фона () в отдельных профилях чипов ChIP для H-NS, IHF и Fis (Grainger et al., 2006). Интересно, что мы также наблюдали очень значительное увеличение связывания Fis в пределах he EPOD (10). Это согласуется с локус-специфической ролью Fis в регуляции генов с высокой экспрессией, включая рибосомные РНК (Aiyar et al., 2002; Cho et al., 2008; Grainger et al., 2006).
снова согласуется с гипотезой о том, что эти регионы представляют собой хромосомные организующие центры (рис. S9). В соответствии с нашими вычислительными анализами выше, мы увидели значительное обогащение для высокоаффинного связывания нуклеоидных белков в наших ts EPOD относительно фона () в отдельных профилях чипов ChIP для H-NS, IHF и Fis (Grainger et al., 2006). Интересно, что мы также наблюдали очень значительное увеличение связывания Fis в пределах he EPOD (10). Это согласуется с локус-специфической ролью Fis в регуляции генов с высокой экспрессией, включая рибосомные РНК (Aiyar et al., 2002; Cho et al., 2008; Grainger et al., 2006).
В сумме наши наблюдения говорят в пользу модели, в которой связывание ts EPOD нуклеоидными белками устанавливает их как центры организации хромосом. Мы утверждаем, что основные биофизические свойства этих областей могут в значительной степени определять эту роль. Известно, что IHF предпочитает изогнутую ДНК, заставляя ее резко изгибаться при связывании; нуклеоидные белки HU и H-NS также прочно связываются с искривленной ДНК (Swinger and Rice, 2004). Fis, H-NS и IHF сдерживают суперспирали (Pettijohn, 1996), а H-NS (Dame et al., 2000) и Fis (Skoko et al., 2006) демонстрируют олигомеризацию и уплотнение ДНК in vitro. Мы предполагаем, что нуклеация начинается с нуклеоидных белков, предпочтительно связывающих эти искривленные области ДНК. Поскольку некоторые нуклеоидные белки предпочитают связываться с искривленной ДНК, эти начальные взаимодействия белок-ДНК делают область более благоприятной для дальнейших событий связывания. Таким образом, волна нуклеоидных белков может распространяться по этим областям, усиливаясь за счет поддержания кривизны и внутридоменных белок-белковых взаимодействий. Гомо- и гетеродимерные белок-белковые взаимодействия, например, как показано для H-NS (Stella et al., 2005), затем могут привести эти домены в контакт друг с другом, образуя классические розеточные структуры, визуализируемые с помощью ЭМ (Delius and Worcel, 19).74а; Постоу и др., 2004).
Fis, H-NS и IHF сдерживают суперспирали (Pettijohn, 1996), а H-NS (Dame et al., 2000) и Fis (Skoko et al., 2006) демонстрируют олигомеризацию и уплотнение ДНК in vitro. Мы предполагаем, что нуклеация начинается с нуклеоидных белков, предпочтительно связывающих эти искривленные области ДНК. Поскольку некоторые нуклеоидные белки предпочитают связываться с искривленной ДНК, эти начальные взаимодействия белок-ДНК делают область более благоприятной для дальнейших событий связывания. Таким образом, волна нуклеоидных белков может распространяться по этим областям, усиливаясь за счет поддержания кривизны и внутридоменных белок-белковых взаимодействий. Гомо- и гетеродимерные белок-белковые взаимодействия, например, как показано для H-NS (Stella et al., 2005), затем могут привести эти домены в контакт друг с другом, образуя классические розеточные структуры, визуализируемые с помощью ЭМ (Delius and Worcel, 19).74а; Постоу и др., 2004).
Наши наблюдения не предполагают, что каждые ts EPOD важны для хромосомной организации во все времена. Скорее, подмножество из ts EPOD может быть вовлечено в формирование структуры более высокого порядка в любой одной клетке или в разных условиях окружающей среды. Соответствующие обсуждения см. в (Deng et al., 2005; Postow et al., 2004; Valens et al., 2004). Отсутствие заметного дефицита приспособленности у штамма E. coli с редуцированным геномом , MDS42 (Колисниченко и др., 2002), у которого отсутствуют 24% ORF, содержащихся в ts EPOD, поддерживает это динамическое и избыточное изображение. Фактически, анализ IPOD этого редуцированного генома показал, что характер занятости остальных EPOD в значительной степени сохраняется, при этом 44% последовательностей EPOD в MDS42 точно перекрываются с последовательностями, определенными в MG1655 (рис. S10). Хотя существует меньшинство локусов с существенно отличающимися паттернами занятости, большая часть остаточных несоответствий связана с различиями в точном определении границ EPOD, а не с их расположением. Эти наблюдения обеспечивают дополнительную поддержку предлагаемой нами модели.
Скорее, подмножество из ts EPOD может быть вовлечено в формирование структуры более высокого порядка в любой одной клетке или в разных условиях окружающей среды. Соответствующие обсуждения см. в (Deng et al., 2005; Postow et al., 2004; Valens et al., 2004). Отсутствие заметного дефицита приспособленности у штамма E. coli с редуцированным геномом , MDS42 (Колисниченко и др., 2002), у которого отсутствуют 24% ORF, содержащихся в ts EPOD, поддерживает это динамическое и избыточное изображение. Фактически, анализ IPOD этого редуцированного генома показал, что характер занятости остальных EPOD в значительной степени сохраняется, при этом 44% последовательностей EPOD в MDS42 точно перекрываются с последовательностями, определенными в MG1655 (рис. S10). Хотя существует меньшинство локусов с существенно отличающимися паттернами занятости, большая часть остаточных несоответствий связана с различиями в точном определении границ EPOD, а не с их расположением. Эти наблюдения обеспечивают дополнительную поддержку предлагаемой нами модели. А именно, что специфические участки хромосом благодаря своему составу последовательностей действуют как расширенные домены занятости белка, что, в свою очередь, может позволить им участвовать в организации крупномасштабной хромосомной топологии. Однако мы также повышаем вероятность того, что установление этих транскрипционно-молчащих белковых доменов занятости может выполнять др. функции. Например, др. приводили доводы в пользу роли нуклеоидных белков, таких как H-NS, в подавлении горизонтально перенесенной ДНК (Dorman, 2007).
А именно, что специфические участки хромосом благодаря своему составу последовательностей действуют как расширенные домены занятости белка, что, в свою очередь, может позволить им участвовать в организации крупномасштабной хромосомной топологии. Однако мы также повышаем вероятность того, что установление этих транскрипционно-молчащих белковых доменов занятости может выполнять др. функции. Например, др. приводили доводы в пользу роли нуклеоидных белков, таких как H-NS, в подавлении горизонтально перенесенной ДНК (Dorman, 2007).
При более внимательном рассмотрении некоторых EPOD можно предположить, что наша автоматическая классификация их на две группы: высокоэкспрессируемые и транскрипционно молчащие, может не охватывать весь спектр их разнообразия. Действительно, один из самых длинных ts EPOD определяется кластером генов, кодирующих ферменты на пути биосинтеза липополисахарида (LPS) (). Анализ содержания специфичной для цепи РНК в этом локусе (рис. S11) ясно показывает, что, хотя этот участок классифицируется как транскрипционно «молчащий», существует низкий уровень экспрессии, который в основном ограничивается первыми тремя генами в опероне (9). 0006 RFAQ , RFAG и RFAP ). Эти наблюдения указывают на то, что расширенная занятость белка может присутствовать в локусах с низким уровнем экспрессии и что это может быть вызвано процессами, отличными от тех, которые действуют в абсолютно молчащих локусах.
0006 RFAQ , RFAG и RFAP ). Эти наблюдения указывают на то, что расширенная занятость белка может присутствовать в локусах с низким уровнем экспрессии и что это может быть вызвано процессами, отличными от тех, которые действуют в абсолютно молчащих локусах.
Мы разработали IPOD, глобальный, in vivo подход для мониторинга занятости белком всего бактериального генома при разрешении отдельных сайтов связывания. Разделение водной и органической фаз ранее использовалось для обогащения на основе плотности нуклеосом в S. cerevisiase (Nagy et al., 2003) и Grainger et al. продемонстрировали, что сшитые последовательности, связанные с РНК-полимеразой, предпочтительно распределяются в органическую фазу в E. coli (Grainger et al., 2006). ). Здесь мы показали, что локализация небольших нуклеопротеиновых комплексов на водной/органической границе является простой, но мощной стратегией для профилирования занятости белка во всем прокариотическом геноме. Хотя идентичность белка, связанного в каждом сайте, неизвестна, все более точные модели специфичности последовательности взаимодействий белок-ДНК должны позволять вероятностные отнесения к известным ДНК-связывающим белкам. Фактически, поскольку анализ IPOD позволяет измерять коррелированную занятость многих сайтов в различных условиях, он должен помочь в уточнении существующих моделей специфичности последовательностей и открытии новых.
Хотя идентичность белка, связанного в каждом сайте, неизвестна, все более точные модели специфичности последовательности взаимодействий белок-ДНК должны позволять вероятностные отнесения к известным ДНК-связывающим белкам. Фактически, поскольку анализ IPOD позволяет измерять коррелированную занятость многих сайтов в различных условиях, он должен помочь в уточнении существующих моделей специфичности последовательностей и открытии новых.
Возможность одновременного мониторинга занятости белком и выхода транскрипции с высоким пространственным и временным разрешением обещает обеспечить истинное системное моделирование динамики транскрипционной сети и хромосомной организации. В больших пространственных масштабах эти данные выявили существование транскрипционно-молчащих белковых доменов занятости. Наши разнообразные наблюдения указывают на то, что эти регионы являются долго предполагаемыми центрами организации доменов хромосомы E. coli .
01
Нажмите здесь для просмотра.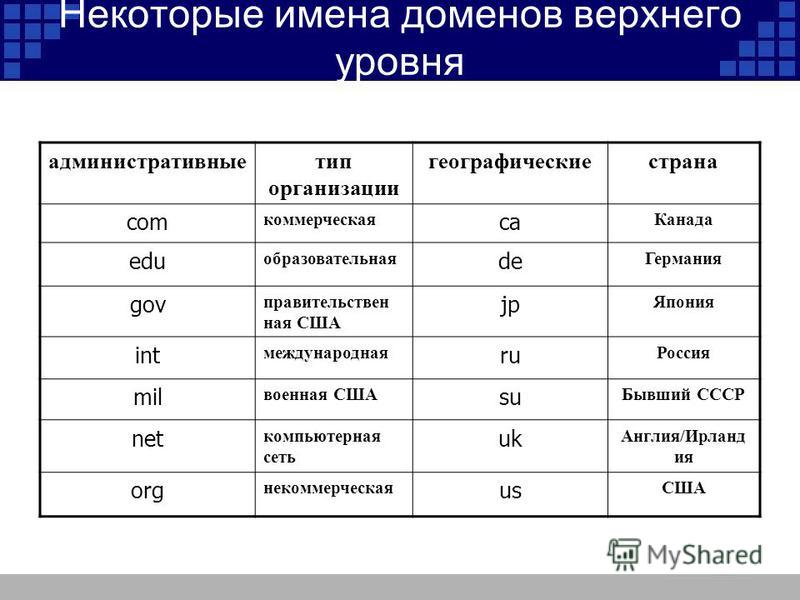 (372K, документ)
(372K, документ)
02
Нажмите здесь для просмотра. (61K, xls)
Мы благодарим сотрудников лаборатории Tavazoie за полезные комментарии к рукописи. Телевидение поддерживалось аспирантской стипендией НАСА. AKH помогла стипендия № 08-1090-CCR-EO Комиссии штата Нью-Джерси по исследованию рака. ST был поддержан грантами NSF (CAREER), DARPA, NHGRI, NIGMS (P50 GM071508) и премией NIH Director’s Pioneer Award (1DP10D003787-01). Данные массива олигонуклеотидов депонированы в NCBI Gene Expression Omnibus под номером доступа {«type»:»entrez-geo»,»attrs»:{«text»:»GSE16414″,»term_id»:»16414″}}GSE16414.
Правовая оговорка издателя: Это PDF-файл неотредактированной рукописи, которая была принята к публикации. В качестве услуги нашим клиентам мы предоставляем эту раннюю версию рукописи. Рукопись будет подвергнута редактированию, набору текста и рецензированию полученного доказательства, прежде чем она будет опубликована в ее окончательной цитируемой форме. Обратите внимание, что в процессе производства могут быть обнаружены ошибки, которые могут повлиять на содержание, и все правовые оговорки, применимые к журналу, относятся к нему.
Обратите внимание, что в процессе производства могут быть обнаружены ошибки, которые могут повлиять на содержание, и все правовые оговорки, применимые к журналу, относятся к нему.
- Айяр С.Э., Маклеод С.М., Росс В., Хирвонен К.А., Томас М.С., Джонсон Р.К., Гурс Р.Л. Архитектура Fis-активируемых транскрипционных комплексов на промоторах rrnB P1 и rrnE P1 Escherichia coli. Дж Мол Биол. 2002; 316: 501–516. [PubMed] [Google Scholar]
- Азам Т.А., Исихама А. Двенадцать видов нуклеоид-ассоциированного белка из Escherichia coli. Специфичность распознавания последовательности и аффинность связывания ДНК. Дж. Биол. Хим. 1999; 274:33105–33113. [PubMed] [Google Scholar]
- Бендич А.Дж. Форма молекул хромосомной ДНК в бактериальных клетках. Биохимия. 2001; 83: 177–186. [PubMed] [Академия Google]
- Чо Б.К., Найт Э.М., Барретт К.Л., Палссон Б.О. Полногеномный анализ связывания Fis в Escherichia coli указывает на причинную роль A-/AT-трактов. Genome Res 2008 [бесплатная статья PMC] [PubMed] [Google Scholar]
- Dame RT, Wyman C, Goosen N.
 Опосредованное H-NS уплотнение ДНК, визуализированное с помощью атомно-силовой микроскопии. Нуклеиновые Кислоты Res. 2000;28:3504–3510. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Опосредованное H-NS уплотнение ДНК, визуализированное с помощью атомно-силовой микроскопии. Нуклеиновые Кислоты Res. 2000;28:3504–3510. [Бесплатная статья PMC] [PubMed] [Google Scholar] - Delius H, Worcel A. Электронно-микроскопические исследования складчатой хромосомы Escherichia coli. Cold Spring Harb Symp Quant Biol. 1974а; 38:53–58. [PubMed] [Google Scholar]
- Delius H, Worcel A. Letter: Электронно-микроскопическая визуализация свернутой хромосомы Escherichia coli. Дж Мол Биол. 1974b; 82: 107–109. [PubMed] [Google Scholar]
- Дэн С., Штейн Р.А., Хиггинс Н.П. Организация доменов суперспиралей и их реорганизация посредством транскрипции. Мол микробиол. 2005; 57: 1511–1521. [Бесплатная статья PMC] [PubMed] [Google Scholar]
- Dorman CJ. H-NS, геномный страж. Nat Rev Microbiol. 2007; 5: 157–161. [PubMed] [Академия Google]
- Гама-Кастро С., Хименес-Хасинто В., Перальта-Хиль М., Сантос-Завалета А., Пеналоза-Спинола М.И., Контрерас-Морейра Б., Сегура-Салазар Х.
 , Мунис-Раскадо Л., Мартинес-Флорес И., Сальгадо Х., и другие. RegulonDB (версия 6.0): модель регуляции генов Escherichia coli K-12 за пределами транскрипции, активные (экспериментальные) аннотированные промоторы и Textpresso-навигация. Нуклеиновые Кислоты Res. 2008; 36:D120–124. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Мунис-Раскадо Л., Мартинес-Флорес И., Сальгадо Х., и другие. RegulonDB (версия 6.0): модель регуляции генов Escherichia coli K-12 за пределами транскрипции, активные (экспериментальные) аннотированные промоторы и Textpresso-навигация. Нуклеиновые Кислоты Res. 2008; 36:D120–124. [Бесплатная статья PMC] [PubMed] [Google Scholar] - Garcia-Russell N, Orchard SS, Segall AM. Исследование структуры нуклеоида у бактерий с использованием хромосомных перестроек, опосредованных интегразой фага лямбда. Методы Энзимол. 2007;421:209–226. [PubMed] [Google Scholar]
- Grainger DC, Hurd D, Goldberg MD, Busby SJ. Ассоциация нуклеоидных белков с кодирующими и некодирующими участками генома Escherichia coli. Нуклеиновые Кислоты Res. 2006; 34:4642–4652. [Бесплатная статья PMC] [PubMed] [Google Scholar]
- Grainger DC, Hurd D, Harrison M, Holdstock J, Busby SJ. Изучение распределения белка цАМФ-рецептора Escherichia coli и РНК-полимеразы вдоль хромосомы E. coli. Proc Natl Acad Sci U S A 2005 [бесплатная статья PMC] [PubMed] [Google Scholar]
- Хиннебуш Б.
 Дж., Бендич А.Дж. Бактериальный нуклеоид визуализируется с помощью флуоресцентной микроскопии клеток, лизированных в агарозе: сравнение Escherichia coli и спирохет рода Borrelia. J Бактериол. 1997;179:2228–2237. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Дж., Бендич А.Дж. Бактериальный нуклеоид визуализируется с помощью флуоресцентной микроскопии клеток, лизированных в агарозе: сравнение Escherichia coli и спирохет рода Borrelia. J Бактериол. 1997;179:2228–2237. [Бесплатная статья PMC] [PubMed] [Google Scholar] - Колисниченко В., Планкетт Г., 3-й, Херринг К.Д., Фехер Т., Посфаи Дж., Блаттнер Ф.Р., Посфаи Г. Разработка уменьшенного генома кишечной палочки. Геном Res. 2002; 12: 640–647. [Бесплатная статья PMC] [PubMed] [Google Scholar]
- Nagy PL, Cleary ML, Brown PO, Lieb JD. Полногеномная демаркация единиц транскрипции РНК-полимеразы II, выявленная путем физического фракционирования хроматина. Proc Natl Acad Sci U S A. 2003;100:6364–6369. [Бесплатная статья PMC] [PubMed] [Google Scholar]
- Pedersen AG, Jensen LJ, Brunak S, Staerfeldt HH, Ussery DW. Структурный атлас ДНК Escherichia coli. Дж Мол Биол. 2000; 299:907–930. [PubMed] [Google Scholar]
- Pettijohn DE. В: Нуклеоид Escherichia coli и Сальмонелла .
 Нейдхардт Ф., редактор. Вашингтон, округ Колумбия: ASM; 1996. С. 158–166. [Google Scholar]
Нейдхардт Ф., редактор. Вашингтон, округ Колумбия: ASM; 1996. С. 158–166. [Google Scholar] - Postow L, Hardy CD, Arsuaga J, Cozzarelli NR. Топологическая доменная структура хромосомы Escherichia coli. Гены Дев. 2004; 18:1766–1779. [Бесплатная статья PMC] [PubMed] [Google Scholar]
- Ren B, Robert F, Wyrick JJ, Aparicio O, Jennings EG, Simon I, Zeitlinger J, Schreiber J, Hannett N, Kanin E, et al. Полногеномное расположение и функция ДНК-связывающих белков. Наука. 2000;290: 2306–2309. [PubMed] [Google Scholar]
- Skoko D, Yoo D, Bai H, Schnurr B, Yan J, McLeod SM, Marko JF, Johnson RC. Механизм сжатия и образования петель хромосом нуклеоидным белком Escherichia coli Fis. Дж Мол Биол. 2006; 364: 777–798. [Бесплатная статья PMC] [PubMed] [Google Scholar]
- Stella S, Spurio R, Falconi M, Pon CL, Gualerzi CO. Природа и механизм олигомеризации нуклеоидного белка H-NS in vivo. Эмбо Дж. 2005; 24: 2896–2905. [Бесплатная статья PMC] [PubMed] [Google Scholar]
- Свингер К.


 check.ru.
check.ru. Опосредованное H-NS уплотнение ДНК, визуализированное с помощью атомно-силовой микроскопии. Нуклеиновые Кислоты Res. 2000;28:3504–3510. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Опосредованное H-NS уплотнение ДНК, визуализированное с помощью атомно-силовой микроскопии. Нуклеиновые Кислоты Res. 2000;28:3504–3510. [Бесплатная статья PMC] [PubMed] [Google Scholar] , Мунис-Раскадо Л., Мартинес-Флорес И., Сальгадо Х., и другие. RegulonDB (версия 6.0): модель регуляции генов Escherichia coli K-12 за пределами транскрипции, активные (экспериментальные) аннотированные промоторы и Textpresso-навигация. Нуклеиновые Кислоты Res. 2008; 36:D120–124. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Мунис-Раскадо Л., Мартинес-Флорес И., Сальгадо Х., и другие. RegulonDB (версия 6.0): модель регуляции генов Escherichia coli K-12 за пределами транскрипции, активные (экспериментальные) аннотированные промоторы и Textpresso-навигация. Нуклеиновые Кислоты Res. 2008; 36:D120–124. [Бесплатная статья PMC] [PubMed] [Google Scholar] Дж., Бендич А.Дж. Бактериальный нуклеоид визуализируется с помощью флуоресцентной микроскопии клеток, лизированных в агарозе: сравнение Escherichia coli и спирохет рода Borrelia. J Бактериол. 1997;179:2228–2237. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Дж., Бендич А.Дж. Бактериальный нуклеоид визуализируется с помощью флуоресцентной микроскопии клеток, лизированных в агарозе: сравнение Escherichia coli и спирохет рода Borrelia. J Бактериол. 1997;179:2228–2237. [Бесплатная статья PMC] [PubMed] [Google Scholar] Нейдхардт Ф., редактор. Вашингтон, округ Колумбия: ASM; 1996. С. 158–166. [Google Scholar]
Нейдхардт Ф., редактор. Вашингтон, округ Колумбия: ASM; 1996. С. 158–166. [Google Scholar]